CRISPR-Cas系统是细菌和古生菌抵抗病毒入侵的一种适应性免疫系统,其中,II型Cas9和V型Cas12系统已被开发为基因编辑工具,广泛应用于基础研究、作物育种、核酸检测、基因治疗等领域1,2。相比于II型Cas9系统,V型Cas12系统具有更加多样性的V-A到V-K核酸内切酶亚型3,其中V-I Cas12 (Cas12i)具有尺寸更小、可自加工为成熟crRNA、可识别TTN PAM序列等特性3,具有体内递送优势。然而,早前发现的Cas12i1和Cas12i2在哺乳动物细胞内编辑活性较低,限制了其应用。
2022年11月15日,《Protein & Cell》期刊在线发表了题为“An engineered xCas12i with high activity, high specificity and broad PAM range”的研究论文,该研究由辉大(上海)生物科技有限公司(简称,辉大基因)研发团队完成。该研究综合利用生信分析、蛋白质工程、流式细胞术、二代测序、PEM-seq和体内外基因治疗有效性评估等技术手段,对辉大基因独立开发的xCas12i进行蛋白工程化改造、筛选及验证,开发出了具有高效靶向编辑活性但极低脱靶编辑活性的高保真xCas12i变体,又称为hfCas12Max。本研究开发的新型DNA编辑工具在基础科学研究、作物育种、核酸检测和基因治疗等领域具有巨大的应用潜力及前景。

在本研究开发中,首先,研究人员利用一种基于单链退火修复(single-strand annealing, SSA)可激活绿色荧光蛋白(EGFP)报告系统,在哺乳动物细胞中检测Cas12i蛋白的靶向编辑活性的提高以及脱靶编辑活性的降低。该可激活荧光报告系统是由分成具有450 bp同源序列的两部分EGFP及中间的一段靶向序列组成(EGxx-target-xxFP),通过靶向切割和SSA激活发出绿色荧光信号。研究人员合成了从宏基因组数据库中找到的10种新型Cas12i,并通过该荧光报告系统筛选到了xCas12i在哺乳动物细胞内具有最高的编辑效率,其荧光强度强于SpCas9和LbCas12a(图1)。
为了进一步提高xCas12i的编辑活性,研究人员利用蛋白质工程手段将xCas12i的PI、RuvC和REC结构域中的氨基酸替换成精氨酸(R),分别通过靶向荧光报告系统和脱靶荧光报告系统筛选靶向编辑活性增强同时脱靶编辑活性降低的突变体。其中,位于PI结构域的N243R和REC结构域的E336R突变显著提高了细胞内靶向编辑活性,位于RuvC切割结构域的D892R突变显著降低了脱靶编辑活性应。通过对大量内源位点编辑效率、报告系统上PAM偏好性以及基于计算机预测或PEM-seq特异性的综合性评估发现,与常见的Cas9和Cas12蛋白相比,突变体xCas12i-N243R/E336R/D892R(hfCas12Max)表现出更宽泛的PAM识别和更高效的编辑效率,且具有相当或更低的脱靶效应(图1)。此外,与脱氨酶融合形成的dCas12Max-ABE和dCas12Max-CBE碱基编辑器也分别表现出较好的A-to-G和C-to-T单碱基编辑效率。
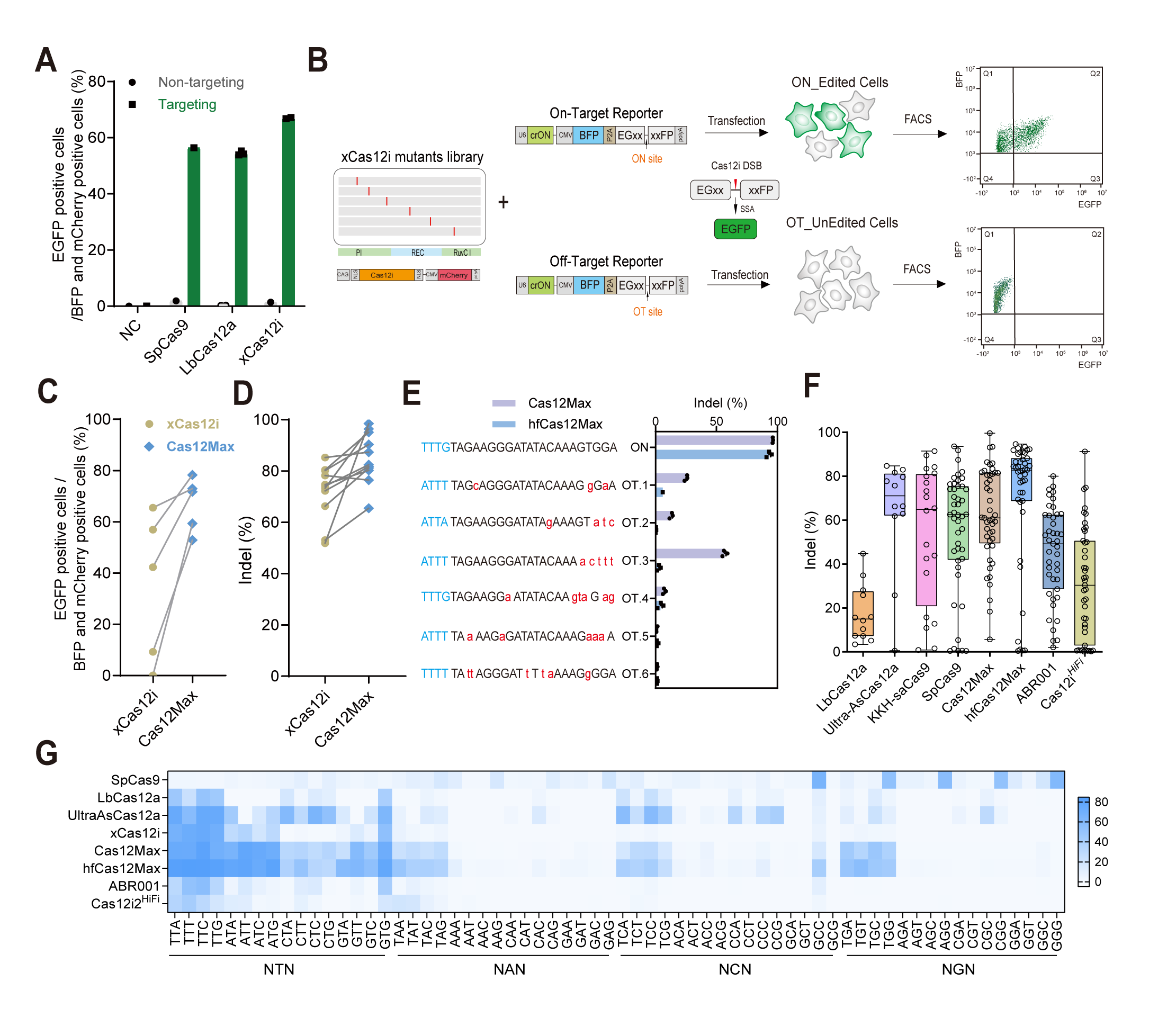
图1:高效高保真xCas12i突变体蛋白的工程化设计、筛选、活性、脱靶效应及PAM识别验证
结合电穿孔递送RNP、靶向二代测序和流式细胞术等技术,研究人员将hfCas12Max RNP递送到CD3+ T细胞并实现高达90%的TRAC基因敲除(图2)。基于脂质纳米颗粒递送系统,研究人员将hfCas12Max的信使RNA和crRNA递送到小鼠体内,并实现接近饱和的Ttr基因敲除(图3)。此外,通过递送dCas12Max-ABE实现接近20%的A-to-G体内编辑效率。综合而言,此项工作开发出了具有高效靶向编辑活性但极低脱靶编辑活性的高保真hfCas12Max及相应的单碱基编辑工具,在体内外基因治疗方面展现了巨大的应用前景。
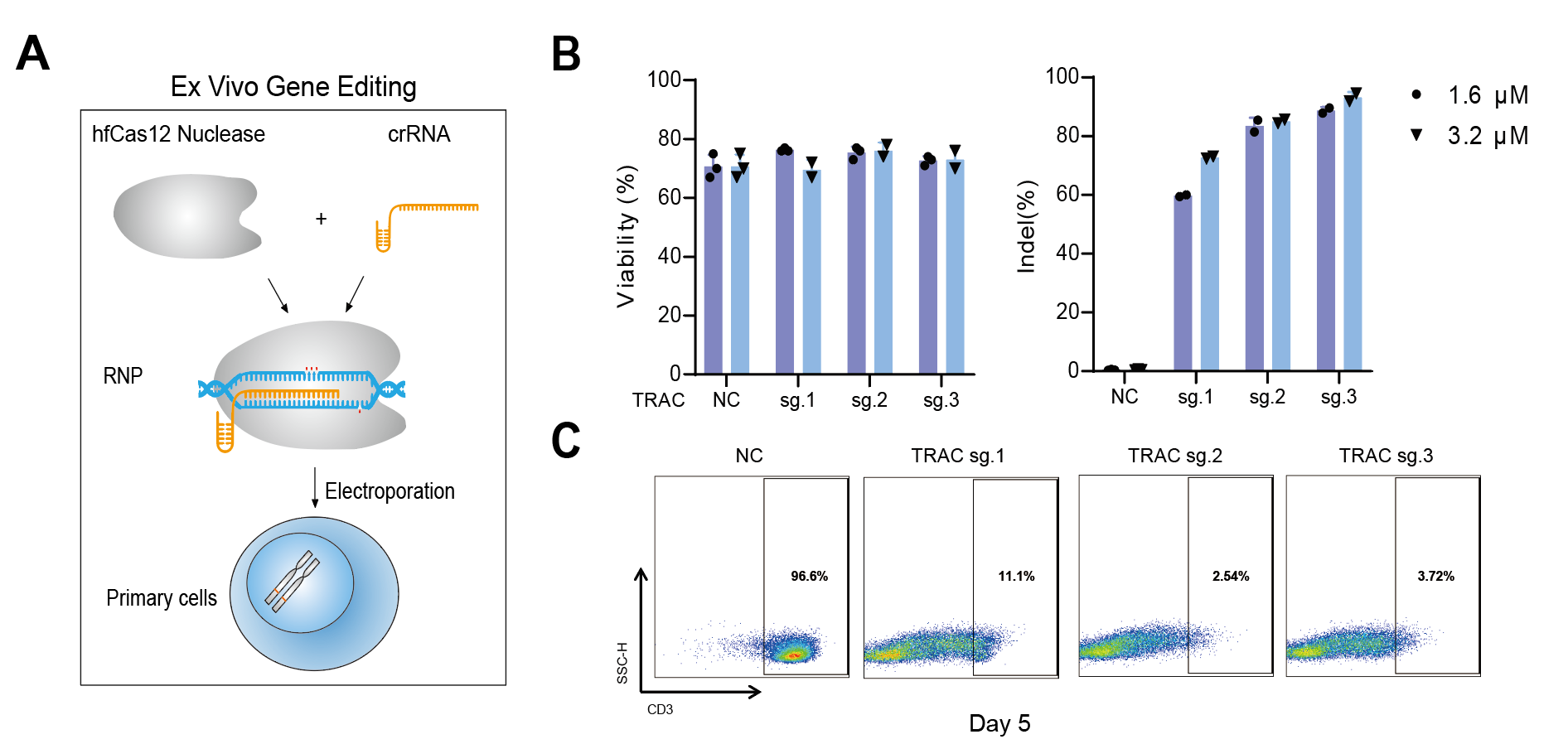
图2:高效高保真hfCas12Max蛋白在CD3+ T细胞中的有效性评估
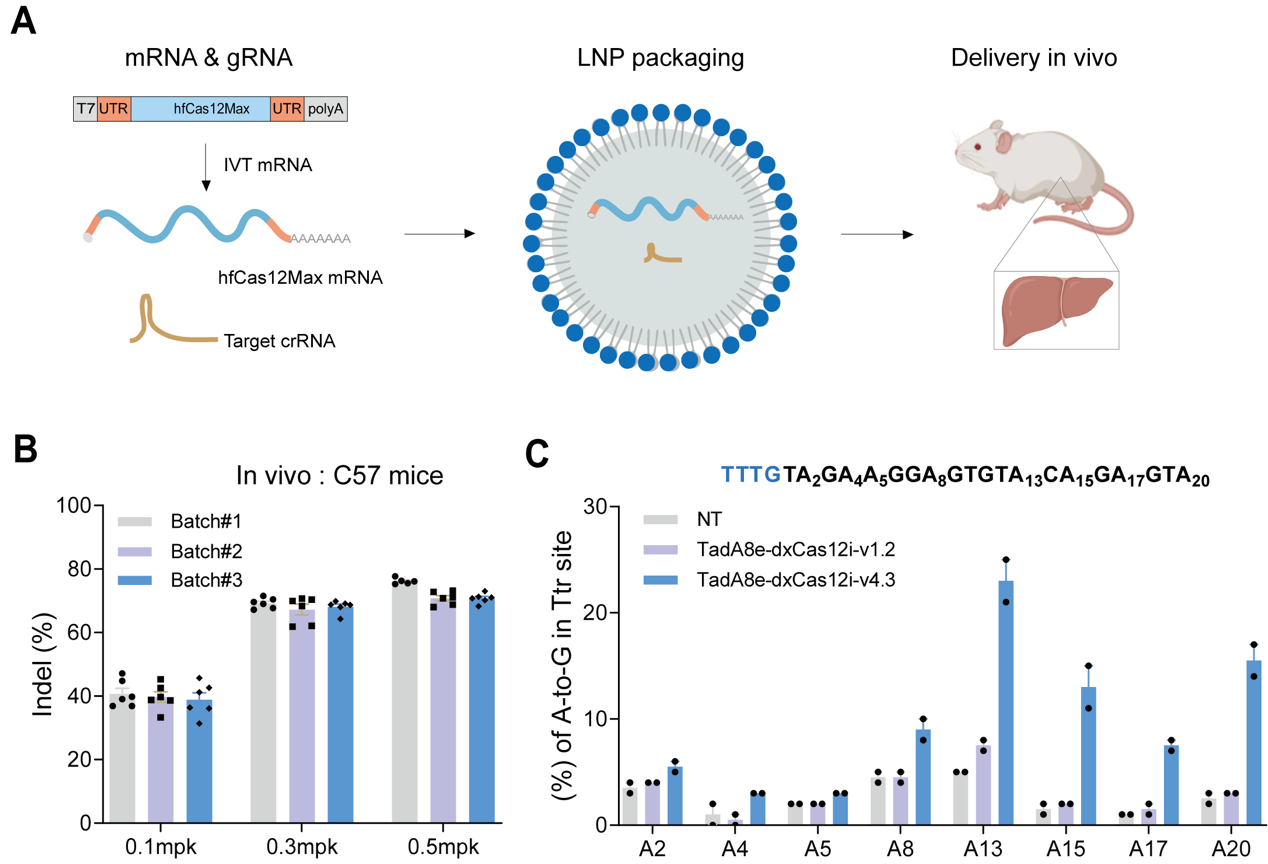
图3:高效高保真hfCas12Max蛋白在哺乳动物细胞及个体中的有效性评估
辉大基因创新研究院张海南、孔祥峰、薛明星博士为本研究论文的共同第一作者,胡静、汪子康、魏迎辉博士等积极参与课题并做出了重要贡献。辉大基因创始人、首席科学顾问杨辉博士和辉大基因创新研究院执行院长周英思博士为该论文的通讯作者。辉大基因已就该项技术提交专利申请。
原文链接:
https://academic.oup.com/proteincell/advance-article/doi/10.1093/procel/pwac052/6827586
参考文献:
1. Anzalone, A. V., Koblan, L. W. & Liu, D. R. Genome editing with CRISPR–Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol 38, 824–844 (2020).
2. Doudna, J. A. The promise and challenge of therapeutic genome editing. Nature 578, 229–236 (2020).
3. Yan, W. X. et al. Functionally diverse type V CRISPR-Cas systems. Science 363, 88–91 (2019).
江苏热线版权及免责声明:
1、凡本网注明 “来源:***(非江苏热线)” 的作品,均转载自其它媒体,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。
2、如因作品内容、版权和其它问题需要同本网联系的,请在30日内进行。